-検索条件
-検索結果
検索 (著者・登録者: qiao & z)の結果414件中、1から50件目までを表示しています

EMDB-43736: 
Umb1 umbrella toxin particle
手法: 単粒子 / : Park YJ, Zhao Q, Seattle Structural Genomics Center for Infectious Disease (SSGCID), DiMaio F, Mougous JD, Veesler D

EMDB-43737: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)
手法: 単粒子 / : Park YJ, Zhao Q, Seattle Structural Genomics Center for Infectious Disease (SSGCID), DiMaio F, Mougous JD, Veesler D

PDB-8w20: 
Umb1 umbrella toxin particle
手法: 単粒子 / : Park YJ, Zhao Q, Seattle Structural Genomics Center for Infectious Disease (SSGCID), DiMaio F, Mougous JD, Veesler D

PDB-8w22: 
Umb1 umbrella toxin particle (local refinement of UmbB1 bound ALF of UmbC1 and UmbA1)
手法: 単粒子 / : Park YJ, Zhao Q, Seattle Structural Genomics Center for Infectious Disease (SSGCID), DiMaio F, Mougous JD, Veesler D

EMDB-37444: 
Cryo-EM structure of PAO1-ImcA with GMPCPP
手法: 単粒子 / : Zhan XL, Zhang K, Wang CC, Fan Q, Tang XJ, Zhang X, Wang K, Fu Y, Liang HH

PDB-8wcn: 
Cryo-EM structure of PAO1-ImcA with GMPCPP
手法: 単粒子 / : Zhan XL, Zhang K, Wang CC, Fan Q, Tang XJ, Zhang X, Wang K, Fu Y, Liang HH

EMDB-35678: 
Apo state of Arabidopsis AZG1 at pH 7.4
手法: 単粒子 / : Xu L, Guo J

EMDB-35679: 
Endogenous substrate adenine bound state of Arabidopsis AZG1 at pH 5.5
手法: 単粒子 / : Xu L, Guo J

EMDB-35680: 
6-BAP bound state of Arabidopsis AZG1
手法: 単粒子 / : Xu L, Guo J

EMDB-35681: 
trans-Zeatin bound state of Arabidopsis AZG1 at pH7.4
手法: 単粒子 / : Xu L, Guo J

EMDB-35682: 
kinetin bound state of Arabidopsis AZG1
手法: 単粒子 / : Xu L, Guo J

EMDB-37658: 
trans-Zeatin bound state of Arabidopsis AZG1 at pH5.5
手法: 単粒子 / : Xu L, Guo J

PDB-8irm: 
Endogenous substrate adenine bound state of Arabidopsis AZG1 at pH 5.5
手法: 単粒子 / : Xu L, Guo J

EMDB-37681: 
Apo state of Arabidopsis AZG1 T440Y
手法: 単粒子 / : Xu L, Guo J

EMDB-35416: 
Cryo-EM structure of the Clr6S (Clr6-HDAC) complex from S. pombe
手法: 単粒子 / : Zhang HQ, Wang X, Wang YN, Liu SM, Zhang Y, Xu K, Ji LT, Kornberg RD

PDB-8ifg: 
Cryo-EM structure of the Clr6S (Clr6-HDAC) complex from S. pombe
手法: 単粒子 / : Zhang HQ, Wang X, Wang YN, Liu SM, Zhang Y, Xu K, Ji LT, Kornberg RD

EMDB-35417: 
Clr6 HDAC (Clr6S)-nucleosome complex
手法: 単粒子 / : Zhang HQ

EMDB-36278: 
Rpd3S-nucleosome (H3K36me3 modified)
手法: 単粒子 / : Wang X, Wang YN, Liu SM, Zhang Y, Xu K, Ji LT, Kornberg RD, Zhang HQ

EMDB-35063: 
The complex structure of Omicron BA.4 RBD with BD604, S309, and S304
手法: 単粒子 / : He QW, Xu ZP, Xie YF

EMDB-35064: 
SARS-CoV-2 Omicron BA.2 RBD complexed with BD-604 and S304 Fab
手法: 単粒子 / : He QW, Xie Y

PDB-8hws: 
The complex structure of Omicron BA.4 RBD with BD604, S309, and S304
手法: 単粒子 / : He QW, Xu ZP, Xie YF
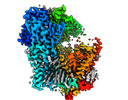
EMDB-36748: 
Structure of a fungal 1,3-beta-glucan synthase
手法: 単粒子 / : Zhao C, You Z, Chen D, Hang J, Wang Z, Meng J, Wang L, Zhao P, Qiao J, Yun C, Bai L

PDB-8jzn: 
Structure of a fungal 1,3-beta-glucan synthase
手法: 単粒子 / : Zhao C, You Z, Chen D, Hang J, Wang Z, Meng J, Wang L, Zhao P, Qiao J, Yun C, Bai L

EMDB-34954: 
Cryo-EM structure of SSX1 bound to the H2AK119Ub nucleosome at a resolution of 3.05 angstrom
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, GuoChao C, Man P, Liu L

EMDB-36747: 
Cryo-EM structure of 2 SSX1 bound to H2AK119Ub nucleosome at a resolution of 3.6 angstroms (2:1 complex)
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, GuoChao C, Man P, Liu L

PDB-8hqy: 
Cryo-EM structure of SSX1 bound to the H2AK119Ub nucleosome at a resolution of 3.05 angstrom
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, GuoChao C, Man P, Liu L
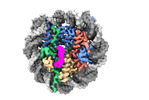
EMDB-34956: 
Cryo-EM structure of SSX1 bound to the unmodified nucleosome at a resolution of 3.02 angstrom
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, Man P, Liu L

EMDB-34957: 
Cryo-EM structure of H2AK119Ub nucleosome at a resolution of 6.11 angstrom
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, Man P, Liu L

PDB-8hr1: 
Cryo-EM structure of SSX1 bound to the unmodified nucleosome at a resolution of 3.02 angstrom
手法: 単粒子 / : Zebin T, Ai HS, Ziyu X, Man P, Liu L

EMDB-34522: 
Cryo-EM Structure of SARS-CoV-2 BA.2 Spike protein in complex with BA7535
手法: 単粒子 / : Liu Z, Yan A, Gao Y

EMDB-34526: 
Cryo-EM structure of SARS-CoV-2 BA.2 RBD in complex with BA7535 fab (local refinement)
手法: 単粒子 / : Liu Z, Yan A, Gao Y

PDB-8h7l: 
Cryo-EM Structure of SARS-CoV-2 BA.2 Spike protein in complex with BA7535
手法: 単粒子 / : Liu Z, Yan A, Gao Y

PDB-8h7z: 
Cryo-EM structure of SARS-CoV-2 BA.2 RBD in complex with BA7535 fab (local refinement)
手法: 単粒子 / : Liu Z, Yan A, Gao Y

EMDB-35790: 
GMPCPP-Alpha1A/Beta2A-microtubule decorated with kinesin
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

EMDB-35791: 
GMPCPP-Alpha1C/Beta2A-microtubule decorated with kinesin
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

EMDB-35792: 
GMPCPP-Alpha4A/Beta2A-microtubule decorated with kinesin
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixa: 
GMPCPP-Alpha1A/Beta2A-microtubule decorated with kinesin non-seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixb: 
GMPCPP-Alpha1A/Beta2A-microtubule decorated with kinesin seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixd: 
GMPCPP-Alpha1C/Beta2A-microtubule decorated with kinesin non-seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixe: 
GMPCPP-Alpha1C/Beta2A-microtubule decorated with kinesin seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixf: 
GMPCPP-Alpha4A/Beta2A-microtubule decorated with kinesin non-seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

PDB-8ixg: 
GMPCPP-Alpha4A/Beta2A-microtubule decorated with kinesin seam region
手法: 単粒子 / : Zheng W, Zhao QY, Diao L, Bao L, Cong Y

EMDB-36579: 
A cryo-EM structure of the bovine chromogranin B dimer at a nominal resolution of ~0.35 nm
手法: 単粒子 / : Jiang QX, Zhu MX, Yadav GP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について










 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
